Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
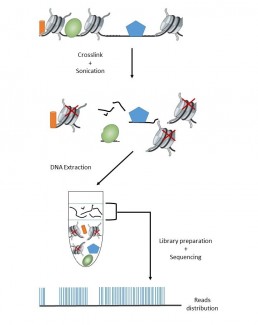
FAIRE seq (Formaldehyde-assisted isolation of regulatory elements) permet d’étudier l’accessibilité de la chromatine par déplétion des nucléosomes. Ces régions de faibles densité en nucléosomes, non liées à des histones, sont identifiées comme régulatrices du génome ( site de fixation de facteurs de transcription , promoteurs ….)
Le principe de la méthode consiste à traiter l’échantillon au formaldéhyde afin de fixer de façon covalente les protéines et l’ADN , préférentiellement les histones plutôt que les facteurs régulateurs du génome. Après fragmentation par sonication, une extraction au Phénol chloroforme permet d’isoler l’ADN « libre » dans la phase aqueuse, les protéines et nucléosomes se retrouvent dans la phase organique.
En pratique , il est bon de prévoir un échantillon contrôle comme référence du génome entier: soit par prélèvement d’un aliquote après fixation qui subirait un « decrosslink » à 65°C soit à partir d’un aliquote qui subirait toute les étapes sauf le traitement au formaldéhyde (Rodrigez-Gil et al. 2018).
Plateformes à contacter pour ce domaine d’expertise
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- Cartographie des sites contacts de la chromatine : CUT & RUN
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie des sites d’accessibilité de la chromatine : DNase-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq