Le séquençage du génome ciblé
Le re-séquençage ciblé permet d’identifier efficacement les variants (SNP, délétions, insertions) pouvant apparaître dans des régions d’intérêt. Cette technique est utilisée particulièrement dans le cas de l’étude d’un panel de gènes associé à une pathologie dans une population ou d’une famille de gènes pour la détermination d’arbres phylogéniques. Il en existe deux approches pour ce séquençage :
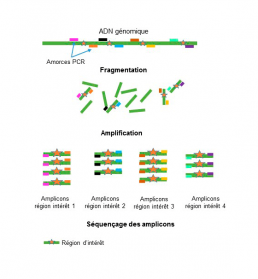
- le séquençage de fragments d’ADN d’intérêt amplifiés par PCR (amplicon) (Fig1).
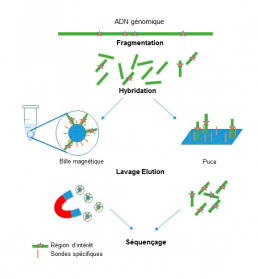
- le séquençage de fragments d’ADN d’intérêt « capturés » par hybridation à des sondes spécifiques des régions à étudier (appâts). On distingue la capture solide de celle en solution. Dans le cas de la capture solide, les sondes sont fixées sur un support solide (puce/array), tandis qu’en capture en solution, celles-ci sont attachées à des billes magnétiques (Fig2).