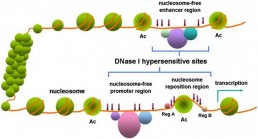
Cartographie des sites d’accessibilité de la chromatine : DNase-seq
La cartographie des régions de chromatine relativement accessible est essentielle pour identifier les éléments de régulation des gènes, y compris les promoteurs et les activateurs. La technique du DNase-seq associe les méthodes de clivage des sites hypersensibles à la Dnase1 (voir fig) et le séquençage à haut débit, permettant ainsi l’identification des régions régulatrices les plus actives pour un type donné de cellules.
Le protocole DNase-seq débute par une préparation de haute qualité des noyaux. Les noyaux sont ensuite incubés avec la DNase 1. Le clivage de l’ADN par l’enzyme doit être optimisé pour éviter une digestion irrégulière ou exagérée de la chromatine. Après avoir dégradé l’ARN et les protéines en utilisant respectivement des RNases et la Protéinase K, les fragments d’ADN sont purifiés et sélectionnés en fonction de la classe de facteurs étudiés (généralement 50-100 pb pour les facteurs de transcription et 130-160 pb pour les nucléosomes). Les fragments isolés sont utilisés pour la construction des banques et le séquençage (Chen et al. 2018).
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- Cartographie des sites contacts de la chromatine : CUT & RUN
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq