Cellule unique
Le séquençage du génome, du transcriptome et de l’épigénome de cellules individualisées a révolutionné de nombreux domaines de la recherche biologique (le développement embryonnaire, la microbiologie, la neurobiologie, l’immunologie, le cancer…). Cette méthode permet, par exemple, d’étudier l’ascendance d’une lignée cellulaire de la cellule souche à la cellule différenciée, ou encore de mettre en évidence une hétérogénéité tissulaire, une modification phénotypique selon le microenvironnement cellulaire, etc. Appliquée en oncologie, elle permet aussi la détection et l’analyse de cellules tumorales circulantes dans le sang, à un stade précoce de la maladie, et facilite le suivi thérapeutique.
Le procédé de séquençage d’une cellule unique comporte plusieurs étapes (revue « Single cell sequencing: a distinct new field » Wang et al. Clin and Translational Med 6 :10 (2017) doi : 10.1186/s40169-017-0139-4) :
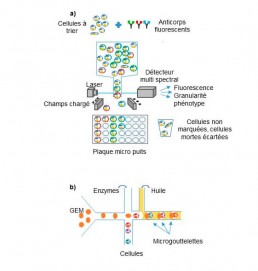
- l’isolement de la cellule : plusieurs méthodes de tri cellulaire sont disponibles selon le tissu ou le bio fluide étudié (FACS; LCM etc.) La micro fluidique tend à supplanter les autres protocoles d’isolement de par son efficacité et son débit de production, bien que le FACS permette de trier selon plusieurs critères phénotypiques (Fig1)
- l’amplification : la quantité de nucléotides extraits d’une cellule (~6 à 10pg) est très insuffisante pour construire les banques en vue du séquençage. Il est donc indispensable d’inclure dans le processus une amplification génomique totale (WGA/WTA :Whole Genome/Trancriptome Ampilfication, MDA : multiple displacement amplification, MALBAC :Multiple annealing and looping based amplification cycles, SMART-seq, etc.)
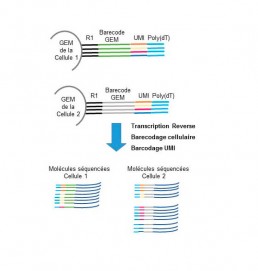
- le barecodage : chaque cellule est identifiée par un barcode unique de même que chaque fragment cDNA amplifié (UMI : Unique Molecular Identifier). La technologie utilisant des GEM (Gel bead in Emulsion) permet ce double barecodage pour le l’étude du transcriptome (Fig2).