scDNA-seq
Selon Evrony GD. et al. (2021) , single-cell DNA-Seq offre un éventail de propriétés qui permettent d’aborder simultanément :
- La détection de mutations somatiques et de CNVs présents à un faible niveau de mosaïsme
- L’évaluation de mosaïsme commun pour la définition de lignage sur un groupe de cellules différenciées
- La détection de particularités de l’ADN associées au phénotype de la cellulaire selon son type et/ou son état biologique
Ainsi, l’approche DNA-Seq sur cellule unique a facilité par exemple
- la compréhension de l’hétérogénéité cellulaire des tumeurs cancéreuses et leur évolution
- la définition de la composition d’un microbiote environnemental, intestinal, etc. et les relations phylogéniques qui en découlent
- la surveillance de pathogènes dans un écosystème
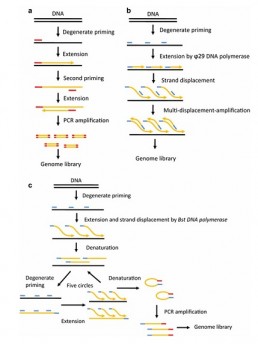
Une limitation importante de cette application réside dans la trop faible quantité d’ADN par cellule pour pouvoir être utilisée sur les séquenceurs disponibles sur le marché. D’où la nécessité d’introduire dans le protocole de préparation des banques une étape d’amplification génomique (WGA), avec le risque d’introduire des erreurs qui pourront perturber les analyses bioinformatiques. Plusieurs méthodes (voir figure) peuvent être utilisées chacune avec leur avantages et défauts ( Wang J and Song Y 2017 )
- DOP-PCR (a): PCR avec des amorces dégénérées pour détecter les CNVs mais pas les SNPs
- MDA (b) : « displacement » amplification pour détecter les SNPs mais avec une couverture hétérogène du génome et des séquences chimères qui polluent les analyses
- MALBAC (c): pré displacement amplification suivie d’une PCR en boucle permettant une bonne fidélité et homogénéité d’amplification sur tout le génome pour la détection de CNVs et SNPs.
Pour appliquer la WGA au haut débit FuY et al. (2019) propose le protocole MICA-eMDA qui introduit la génération d’une émulsion ou chaque gouttelette est le siège d’une pré amplification.
Plateformes à contacter pour ce domaine d’expertise