Cartographie des marques épigénétiques de l’ADN : Methyl seq
La méthylation de l’ADN est une modification chimique post-réplication des cytosines (5mC) au niveau des loci CpG. L’état de méthylation de l’ADN est variable le long du génome et peut modifier de façon significative l’expression de certains gènes et affecter le remodelage de la chromatine.
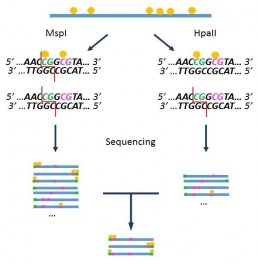
« Methyl Seq » est une méthode d’évaluation de la méthylation de l’ADN qui exploite l’activité différentielle des enzymes de restriction MspI et HpaII en fonction de l’état de méthylation de leurs sites de restriction (AL Brunner et al). Même si elle présente quelques limitations (ne détecte que les sites CpG incluent dans les sites de restrictions), cette méthode donne un reflet de la méthylation globale du génome.
L’échantillon est séparé en 2 aliquotes, l’un incubé avec MspI qui digère le site de restriction indépendamment de l’état de méthylation des cytosines, l’autre avec HpaII qui ne digère que les sites non méthylés. Après réparations des extrémités, les fragments sont indexés avec les adaptateurs requis pour le séquençage NGS.
L’analyse comparative des données de séquençage permet d’identifier les séquences dérivées de sites méthylés.
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- BiSeq