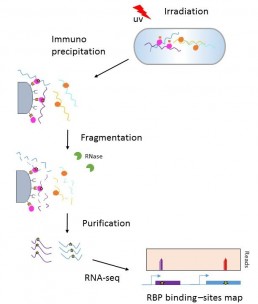
Cartographie des sites des interactions ARN- protéines : CLIP seq
L’analyse des interactions ARN-protéines permet de mieux comprendre le devenir post transcriptionnel des ARNs (leur stabilité, leur localisation , leur transport, leur niveau/efficacité de traduction etc. ). CLIP Seq (Crosss-Linking ImmunoPrecipitation Sequencing) est une méthode utilisée pour l’identification des sites cibles pour la liaison des protéines (RBP) sur l’ARN. Cette technologie combine l’immunoprécipitation de RBPs d’intérêt et le séquençage haut débit de l’ARN.
Le principe de la méthode consiste à fixer de façon covalente par irradiation aux ultra violets ou réaction enzymatique, les protéines RBPs à l’ARN. Après lyse cellulaire, les complexes ARN-protéines sont isolés par immunoprécipitation en présence des anticorps d’intérêt. L’étape suivante consiste à éliminer les fragments libres d’ARN par digestion par une RNase. Après ligation des adaptateurs, élimination des protéines et purification , les sites d’interaction avec les RBPS sont séquencés par RNAseq.
Ce protocole complexe est détaillé dans C.Y. Flora et al. 2018
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq