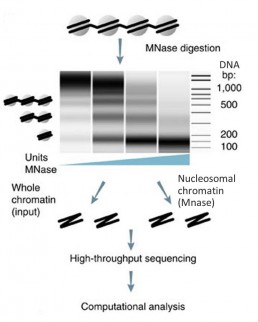
Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
L’agencement des nucléosomes est un aspect important de l’organisation de la chromatine, avec des implications directes sur la régulation des gènes. La technique usuelle de cartographie des nucléosomes est basée sur la digestion par la nucléase micrococcale (MNase) qui dégrade préférentiellement l’ADN non protégé par des protéines. Par comparaison avec un échantillon contrôle n’ayant pas subit la digestion MNAse, on peut indirectement déterminer les sites d’accessibilité de la chromatine.
Pour éviter toute re -conformation de la chromatine lors de son extraction ou son morcellement, il est recommandé de la figer par traitement au formaldéhyde. L’étape de digestion enzymatique à la MNase est un point délicat de la méthode, car les concentrations et les temps d’incubation doivent être choisi de façon à isoler toutes les classes de nucléosmes et d’obtenir des fragment d’ADN mononucléosomal de 200bp à 500 bp sans pour autant aboutir à la détérioration de l’ADN (voir figure). Les complexes ADN – histones sont ensuite disjoints et l’ADN purifié peux intégrer le protocole de séquençage (Mieczkowski et al. 2016).
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq