RAD-Seq
RAD seq (Restriction‐site Associated DNA sequencing) représente une alternative au séquençage NGS du génome entier pour la détection, sans a priori, de variations SNPs et indels de l’ADN. Cette méthode consiste dans le séquençage ciblé des régions adjacentes à des sites de restriction communs repartis sur tout le génome. L’utilisation de plusieurs enzymes de restriction augmentera la couverture du génome, sans qu’un génome de référence soit nécessaire.
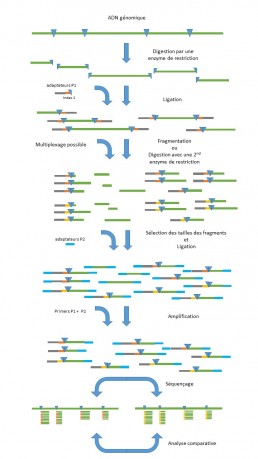
Après digestion par une enzyme de restriction, une première ligation permet d’ajouter aux fragments d’ADN un adaptateur P1, complémentaire au site de restriction et barcodé pour l’identification des échantillons. P1 contient aussi les sites des amorces d’amplification et de séquençage. L’étape suivante est une fragmentation mécanique associée à une sélection de la taille des fragments selon les critères du protocole de séquençage. Pour un meilleur ajustement de l’étape de sélection de la taille, la fragmentation peut être remplacée par la digestion avec une seconde enzyme de restriction (ddRADseq). L’adaptateur P2 est ensuite ligué aux fragments isolés. Une PCR avec les amorces d’amplification P1 et P2 permettra d’amplifier uniquement les fragments portant les adaptateurs P1 et P2 à leurs extrémités (cf Figure).
RADseq est un outil puissant de la génomique des populations applicable aux analyses comparatives (Perry EB et al. 2017) et évolutives dans le contexte d’organismes qui n’ont pas de génome de référence disponible (Andrews et al. 2016).
Plateformes à contacter pour ce domaine d’expertise